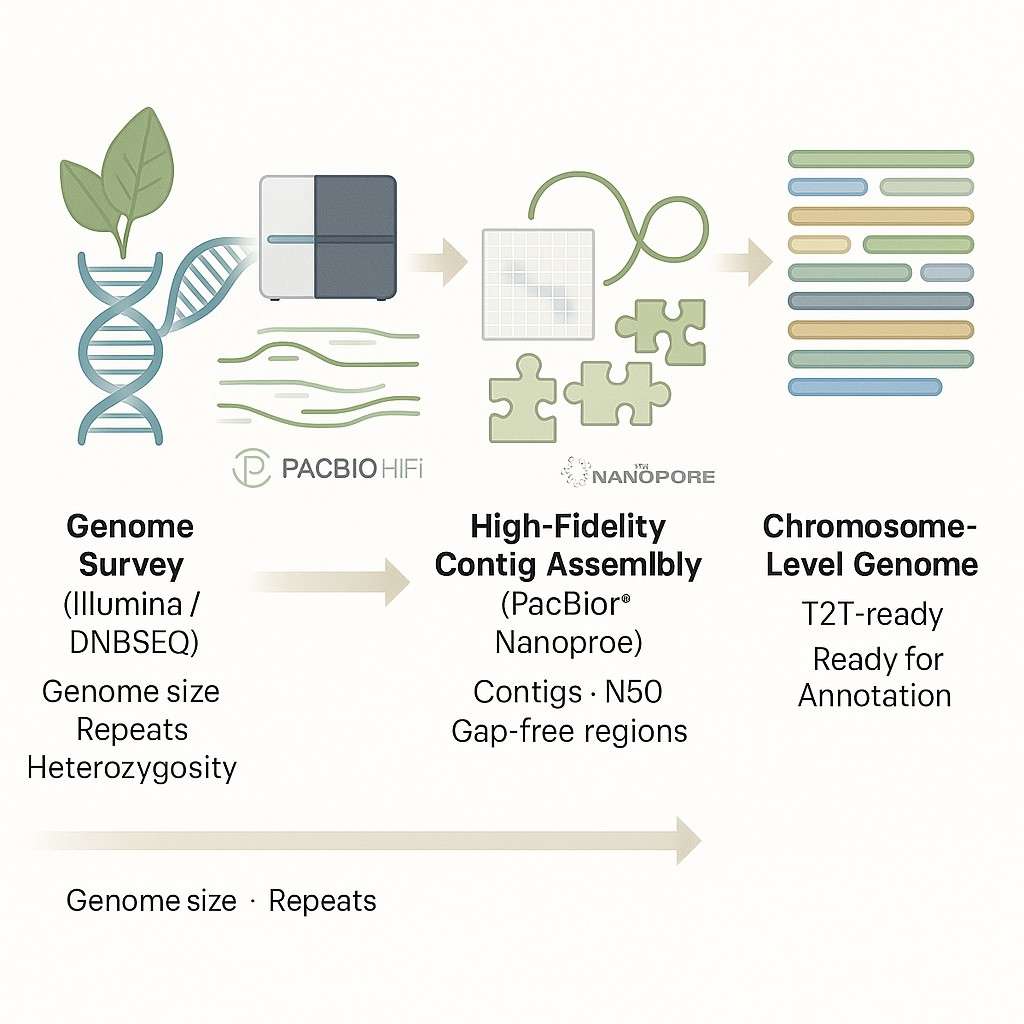
Was ist die de novo Sequenzierung des gesamten Genoms von Pflanzen und Tieren?
Pflanzen- und Tiergenom-De-novo-Sequenzierung bezieht sich auf das Zusammenstellen eines vollständigen Genoms, ohne auf eine vorhandene Referenzsequenz zurückzugreifen. Dieser Ansatz ist entscheidend, wenn man mit Arten arbeitet, die über kein Referenzgenom verfügen, schlecht assemblierte Genome haben oder komplexe genomische Merkmale wie hohe Heterozygotie, Polyploidie oder umfangreiche repetitive Regionen aufweisen.
Anstatt Sequenzierungsreads an ein bekanntes Genom auszurichten, rekonstruiert die de-novo-Assemblierung das Genom von Grund auf neu – ähnlich wie das Lösen eines riesigen Puzzles mit nur den Sequenzfragmenten, die von Hochdurchsatz-Sequenzierungsplattformen erzeugt werden. Das Ergebnis ist ein hochauflösendes genetisches Blueprint, das für funktionale Annotation, vergleichende Analysen, molekulares Züchten und evolutionäre Studien verwendet werden kann.
Bei CD Genomics bieten wir umfassende de novo Sequenzierungsdienste für eine Vielzahl von Pflanzen- und Tierarten an. Durch die Integration von Illumina-Kurzlesungen, PacBio HiFi-Lesungen, Nanopore-Ultra-Lesungen und Hi-C-Chromatin-Interaktionsdaten liefern wir Chromosomenebene Genomassemblierungen bereit für nachgelagerte Forschung und Veröffentlichung.
Wann sollten Sie De-Novo-Genomsequenzierung verwenden?
De-novo-Genomsequenzierung ist die bevorzugte Strategie, wenn kein hochqualitatives Referenzgenom vorhanden ist oder wenn bestehende Referenzen Ihre Forschungsziele nicht erfüllen können. Hier sind die häufigsten Anwendungsfälle:
✅ Kein Referenzgenom verfügbar
Für neu entdeckte oder wenig erforschte Arten ermöglicht die de-novo-Sequenzierung Forschern, eine vollständige Referenz von Grund auf zu erstellen.
✅ Unvollständige oder fragmentierte Referenz
Viele öffentlich verfügbare Genome sind veraltet, schlecht assembliert oder auf Scaffold-Ebene fragmentiert. De-novo-Assemblierung liefert Chromosomenebene Kontinuität für hochauflösende Forschung.
✅ Komplexe Genome: Polyploidie, Heterozygotie, Wiederholungen
Pflanzen- und Tiergenome enthalten häufig hohe Mengen an Duplikationen, strukturellen Variationen oder repetitiven Elementen. De-novo-Ansätze, die verwenden Langzeit-Sequenzierung und Hi-C-Kartierung diese Herausforderungen überwinden.
✅ Pan-Genom-Konstruktion
Wenn ein einzelnes Referenzgenom die genetische Vielfalt einer Art nicht erfassen kann, zeigt der Aufbau eines Pan-Genoms durch de-novo-Assemblierung mehrerer Individuen populationsspezifische Variationen.
✅ Merkmalsentdeckung und molekulare Züchtung
Hochwertige Baugruppen bilden die Grundlage für GWAS, QTL-Kartierungund Genom-Editierung—insbesondere in der Landwirtschaft, Aquakultur und Tierforschung.
Profi-Tipp:
De-novo-Sequenzierung ist nicht nur für neuartige Arten. Sie ist oft die beste Möglichkeit zur Aufrüstung ein niedrig-kontigiertes Genom in veröffentlichungsreife Qualität, insbesondere in Kombination mit HiFi- und Hi-C-Daten.
Technologie-Strategieübersicht: Plattformvergleich für die Genomassemblierung
| Plattform | Rolle in der Versammlung | Typische Abdeckung | Stärken | Empfohlen für |
|---|---|---|---|---|
| Illumina / DNBSEQ™ | Genomüberprüfung, Fehlerkorrektur | 30–50× | Hohe Genauigkeit, niedrige Kosten, essenziell für k-mer-Profiling | Erste Analyse der Genomkomplexität |
| PacBio HiFi | Contig-Ebene de novo Assemblierung | 30–60× | Ultra-hohe Genauigkeit (Q20+), hervorragend für wiederholungsreiche oder polyploide Genome | Pflanzen-/Tiergenome mit hoher Heterozygotie |
| Oxford Nanopore (ONT) | Lückenfüllung, ultra-lange Leseassemblierung | 50–100× | Ultra-lange Reads (>100 kb), ideal für Telomer-zu-Telomer (T2T) Assemblierungen | Genome, die vollständige oder nahezu vollständige Kontinuität erfordern |
| Hi-C | Chromosomen-skalige Gerüstbildung | 100–150× | Erstellt Chromosomen-Pseudomoleküle, korrigiert Fehlassemblierungen | Endgültige Chromosomenverankerung und Qualitätskontrolle |
| 10x Genomics Linked-Reads | Wiederholungsauflösung, Phasung (optional) | ~60× | Phasen heterozygoter Loci unterstützen die Haplotyp-Trennung. | Diploide oder hoch heterozygote Arten |
| BioNano-Optische Kartierung | Erkennung großer struktureller Variationen (optional) | NA | Erkennt SVs, erstellt komplexe Assemblierungen | Sehr große oder strukturell komplexe Genome |
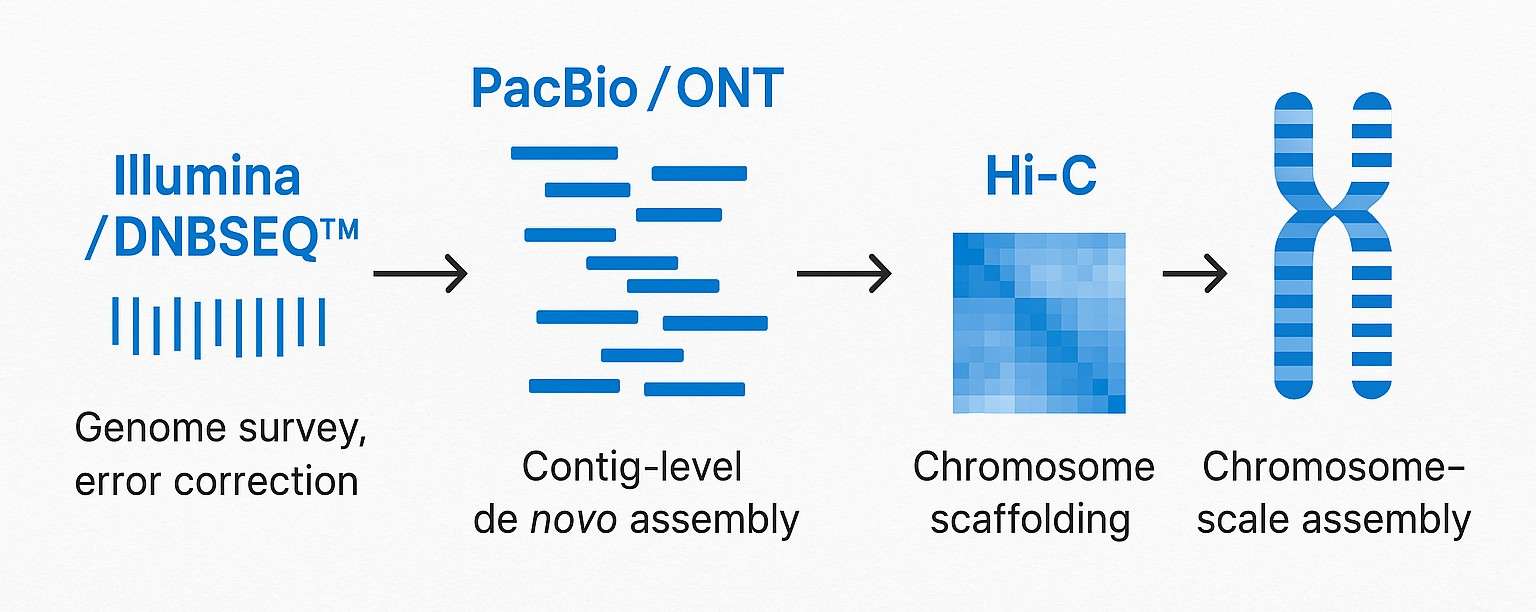 Hybrid-Strategie-Einsicht:
Hybrid-Strategie-Einsicht:
Die erfolgreichsten Assemblierungen kombinieren kurze Reads + lange Reads + Hi-C. Wir passen die Plattformkombination basierend auf Genomgröße, Ploidie und Ihren Forschungszielen an.
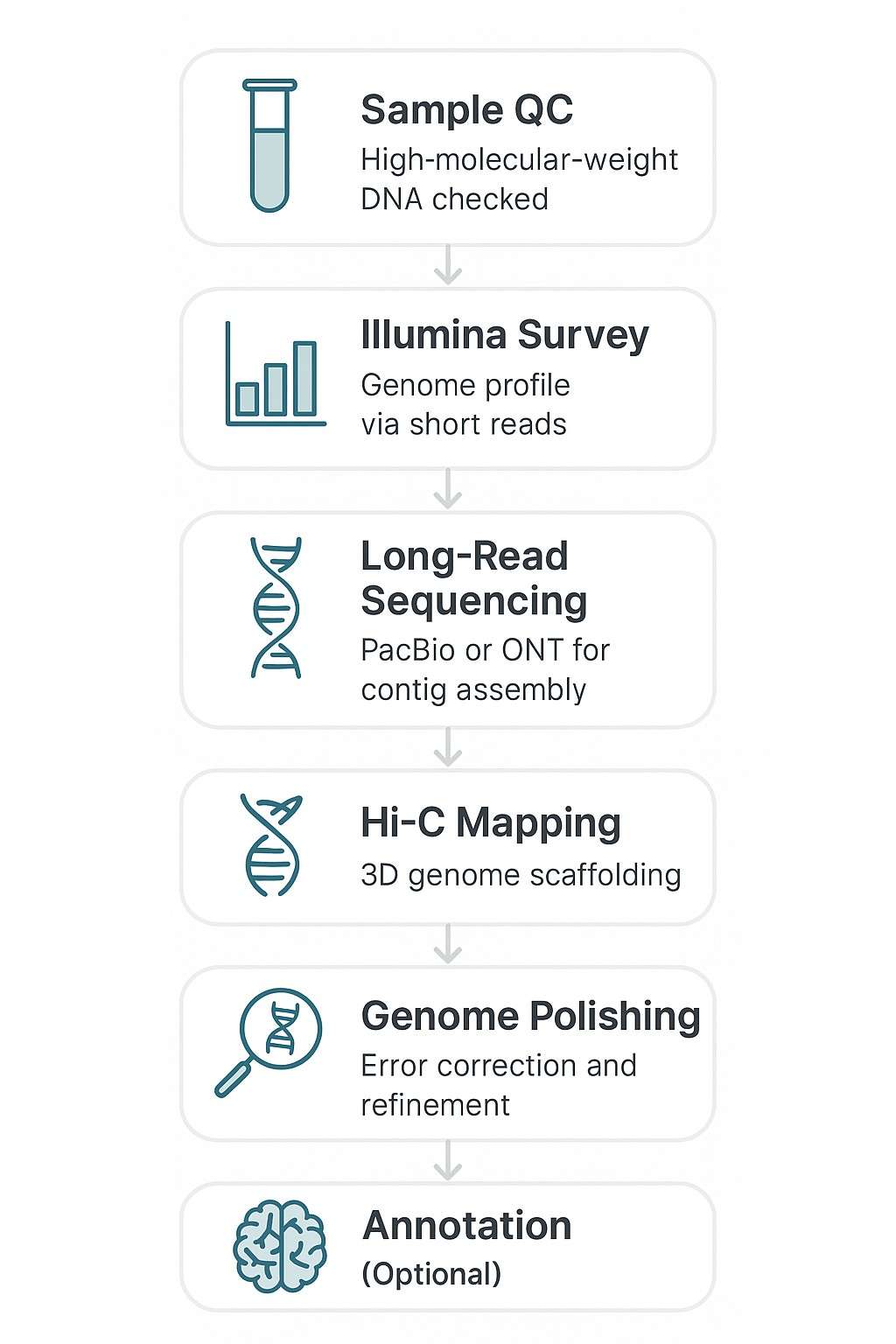
De Novo Genomsequenzierungsdienst-Workflow: Vom Sample zur Chromosomen-großen Assemblierung
Musterqualitätskontrolle
- Integritätsbewertung über PFGE oder Femto Pulse
- Reinheitsprüfungen (OD-Verhältnisse, Qubit und Entfernung von RNA-Kontamination)
Genomüberprüfung (Illumina)
- Kurzzeit-Sequenzierung (~100X Abdeckung)
- K-mer-Analyse für Genomgröße, Wiederholungsinhalt, Heterozygotie
- Leitfäden für die Downstream-Long-Read- und Hi-C-Strategie
Langzeit-Sequenzierung (PacBio HiFi oder Oxford Nanopore)
- Hochkontinuierliche de novo Assemblierung von primären Contigs
- Plattformen ausgewählt basierend auf den Eigenschaften des Zielgenoms
- 30–100X Abdeckung je nach Plattform
Gerüstbildung und Chromosomenverankerung (Hi-C-Sequenzierung)
- Erfasst langreichweitige Chromatin-Interaktionen
- Verankert Contigs an Pseudochromosomen
- Ermöglicht die Chromosomen-große Genomassemblierung
Polieren und Fehlerkorrektur
- Kurzlese-Polierung zur SNP-/Indel-Korrektur
- Lückenfüllung und Wiederholungsauflösung
- BUSCO und qualitätsbasierte Überprüfungen auf Basis von Alignments
Genomannotation (optionale Ergänzung)
- Vorhersage der Genstruktur (ab initio und evidenzbasiert)
- Wiederholte Regionsmaskierung
- Funktionale Annotation (GO, KEGG, Pfam)
Bioinformatikanalyse
Unsere Genom-Informationspipeline integriert Hochdurchsatz-Assemblierung, Annotation und vergleichende Analyse – maßgeschneidert für Pflanzen- und Tierarten. Egal, ob Sie mit einem diploiden, polyploiden oder hochrepetitiven Genom arbeiten, wir bieten skalierbare und präzise Lösungen zur Entschlüsselung von Komplexität.
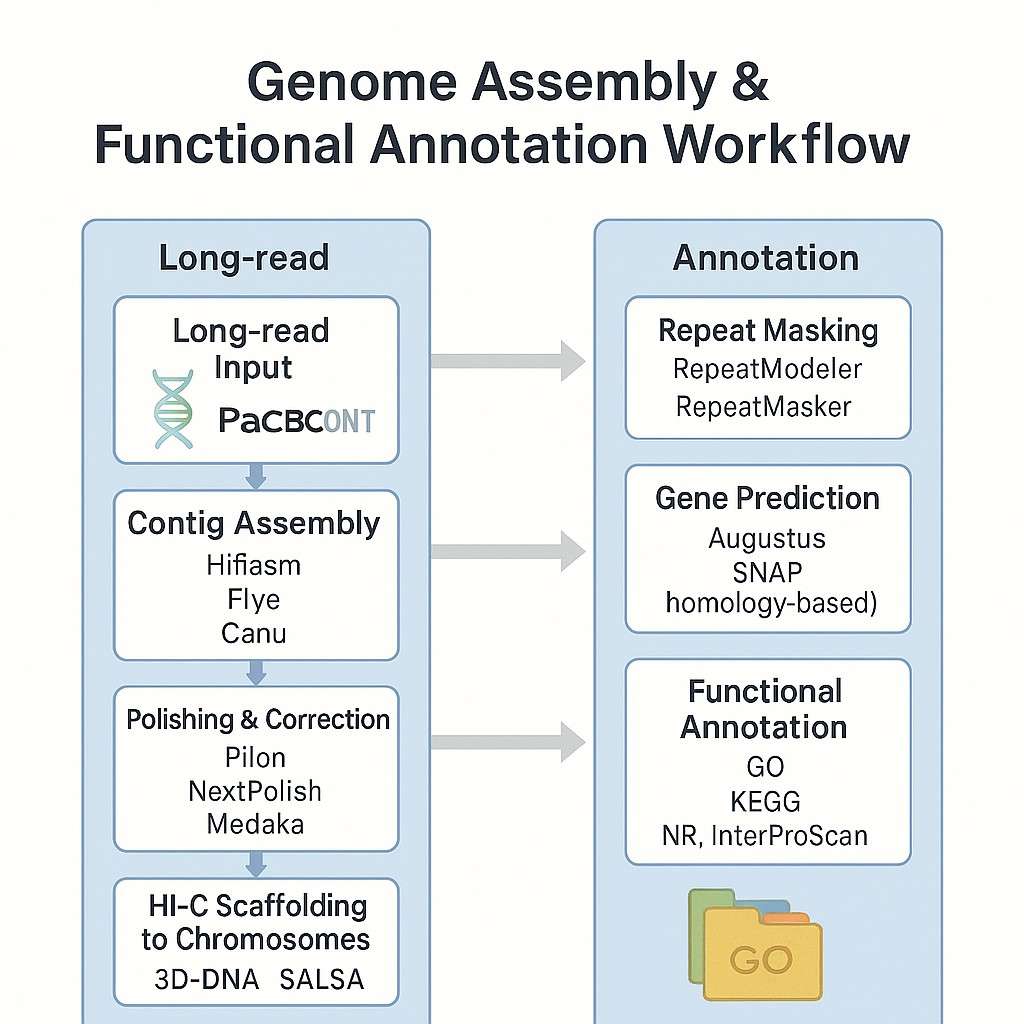
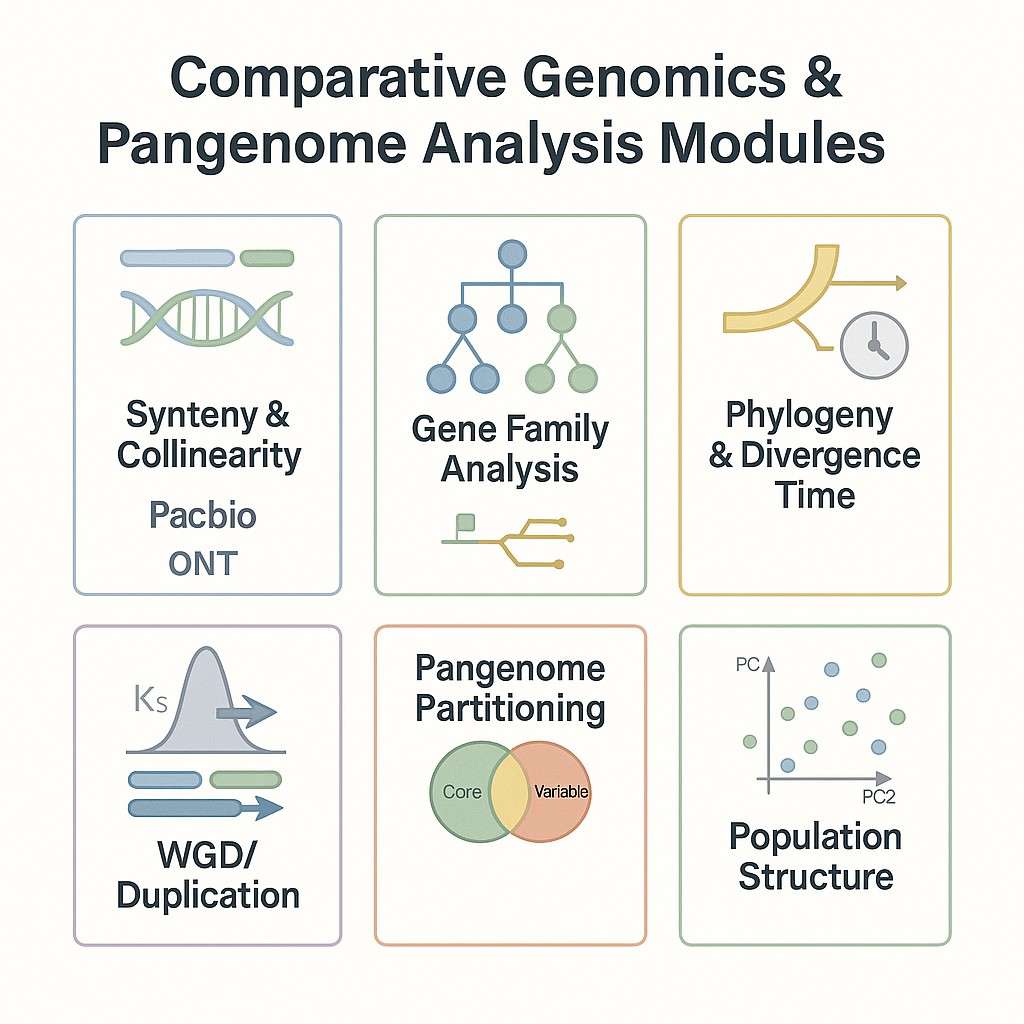
Arbeitsablauf

Musteranforderungen & Qualitätsstandards
| Probenart | Erforderlicher Betrag | Reinheitskriterien | Besondere Hinweise |
|---|---|---|---|
| Frisches oder gefrorenes Tiergewebe | ≥ 1,5 μg gDNA (≥50 kb durchschnittliche Länge) | OD260/280: 1,8–2,0; OD260/230: ≥2,0 | Vermeiden Sie blutkontaminierte Proben; keine Frost-Tau-Zyklen. |
| Pflanzenblätter oder -stängel | ≥ 2 μg gDNA (≥50 kb durchschnittliche Länge) | Gleich wie oben | Vermeiden Sie Kontaminationen mit Polysacchariden und Polyphenolen; bevorzugen Sie junge, zarte Gewebe. |
| Kultivierte Zellen (z. B. Fische, Insekten) | ≥ 1,5 μg gDNA | Das Gleiche wie oben | Für Insekten das Chitin-Exoskelett vor der Extraktion entfernen. |
| Hi-C quervernetztes Gewebe | ≥ 1 g frisches Gewebe oder ~5 Millionen Zellen | OD nicht anwendbar (vernetzt) | Kreuzvernetzung und Fixierung müssen unserem Hi-C-Vorbereitungsprotokoll folgen. |
Allgemeine QC-Kriterien:
- Hochmolekulare DNA: >50 kb bevorzugt für Langlese-Plattformen (PacBio HiFi, Oxford Nanopore)
- Keine Kontamination mit RNA, Proteinen oder sekundären Metaboliten
- Konzentration: ≥50 ng/μL (Qubit); Integrität: Bestätigt durch Pulsfeldgel oder Femto Pulse
Brauchen Sie Hilfe bei der DNA-Extraktion?
CD Genomics bietet umfassende Extraktionsdienste, die auf Pflanzen- und Tiergenome zugeschnitten sind, und verwendet zur Minimierung von Scherung und Verunreinigungen die Magnetperlenreinigung. Kontaktieren Sie uns, um mehr zu erfahren.
Liefergegenstände
CD Genomics bietet umfassende und gut organisierte Ergebnisse für jedes Pflanzen- oder Tier-Whole-Genome-De-Novo-Sequenzierungsprojekt. Unsere Datenpakete sind auf eine nahtlose nachgelagerte Analyse und Publikationsbereitschaft zugeschnitten.
✅ Standard-Lieferungen
| Dateityp / Inhalt | Beschreibung |
|---|---|
| Rohsequenzierungsdaten | FASTQ-Dateien von PacBio HiFi, Nanopore, Illumina und/oder Hi-C-Plattformen |
| Versammlungsresultate | Genom-Contigs und -Scaffolds im FASTA-Format |
| Versammlungsmetrikenbericht | Zusammenfassung der Genomgröße, N50, GC-Gehalt, Vollständigkeit (BUSCO usw.) |
| Genomannotation (Optional) | GFF3/GTF-Dateien, funktionale Annotationsdaten, Visualisierung der Genstruktur |
| Hi-C Interaktionskarte | Kontaktmatrizen und Assemblierungsgerüstdiagramme (falls Hi-C enthalten ist) |
| Circos- und Synteny-Diagramme | Visuelle Zusammenfassungen der Genomarchitektur und vergleichende Analysen |
| Bioinformatik Zusammenfassungsbericht | Detaillierte Methoden, Softwareversionen und Pipeline-Beschreibungen |
✅ Optionale Zusatzleistungen (Projekt-Upgrades)
Für Projekte, die eine fortgeschrittene Datenanalyse oder maßgeschneiderte Ergebnisse erfordern, bietet CD Genomics die folgenden Upgrade-Optionen an:
| Upgrade-Option | Beschreibung |
|---|---|
| Chromosomenebene Assemblierung | Erreicht durch Hi-C oder BioNano-Scaffolding, das chromosomale Pseudomoleküle liefert. |
| Funktionale Genomannotation | Beinhaltet Genvorhersage, GO/KEGG-Anreicherung, Wiederholungselemente und TE-Anmerkungen. |
| Vergleichende Genomik-Paket | Umfasst die gesamte Genom-Syntenie, Ortholog-Clusterbildung und Schätzung der evolutionären Distanz. |
| Pan-Genom-Konstruktion | Multi-Proben-Assemblierung-Integration, strukturelle Varianten-Erkennung und gemeinsame/einzigartige Gen-Sets |
| Epigenom-Integration | Add-On für Methylierungs- oder Histonmodifikationskarten (benötigt kompatible Probenvorbereitung) |
| GWAS-bereite Datenformatierung | Beinhaltet SNP/INDEL-Calls, VCF-Formatierung und Dateien zur Populationsstruktur für GWAS-Pipelines. |
Hervorgehobenes Projektübersicht
| Artenart | Genomgröße | Contig-Anzahl | Contig N50 | Hi-C Verankerungsrate |
|---|---|---|---|---|
| Pflanze A | 1,02 Gb | 626 | 7,15 MB | 95,4 % |
| Pflanze B | 793,46 MB | 347 | 34,19 MB | 96,1 % |
| Wassertier A | 979,98 MB | 513 | 5,36 MB | 97,89% |
| Wassertier B | 827,62 MB | 170 | 9,88 MB | 99,51 % |
| Säugetier | 3,3 Gb | 2.658 | 79,41 MB | 98,58 % |
| Insekt | 979,98 MB | 513 | 5,37 MB | 97,89% |
Diese hochkontinuierlichen Genome zeigen die robuste Assemblierungs-Pipeline von CD Genomics über verschiedene Arten hinweg – von komplexen Pflanzengenomen bis hin zu Chromosomenebene-Assemblierungen bei Säugetieren und aquatischen Organismen.
Teilweise Ergebnisse sind unten aufgeführt:
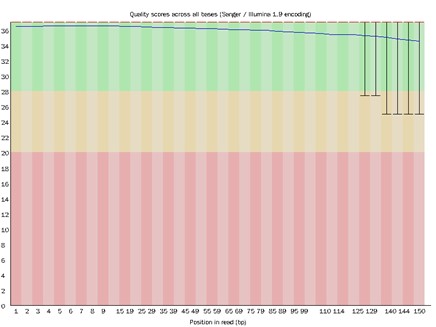
Verteilung der Basisqualität.
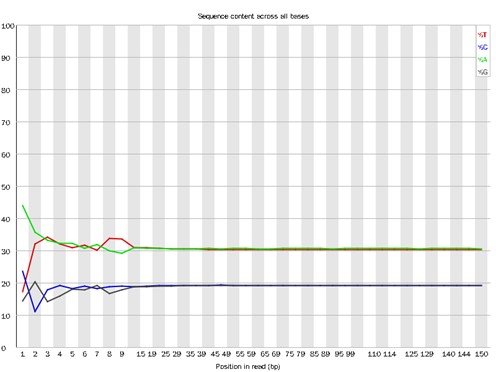
Verteilung des Basisinhalts.
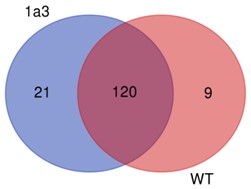
Geteilte SNP-Nummer zwischen Proben.
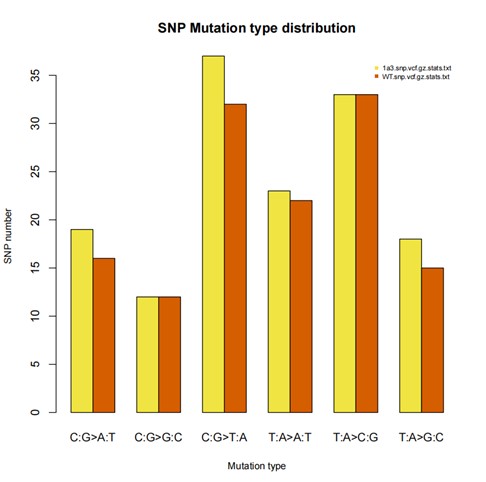
Verteilung der SNP-Mutationsarten.
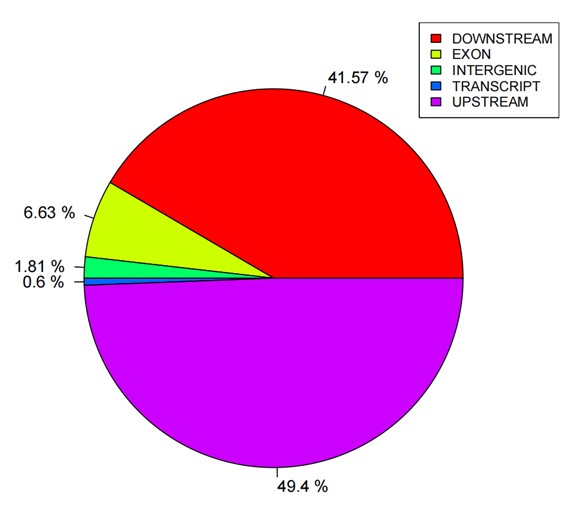
Statistikdiagramm der SNP-Anmerkungen.
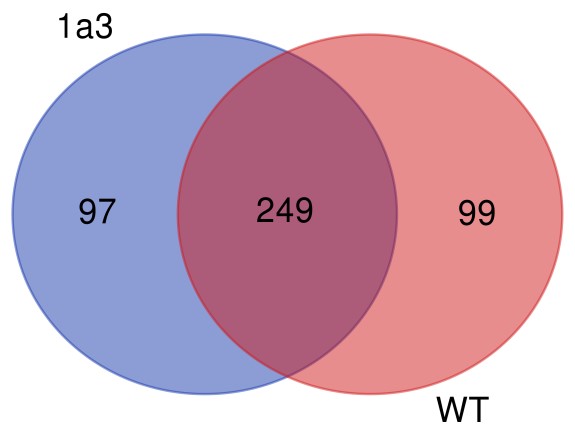
Geteilte InDel-Zahl zwischen Proben.
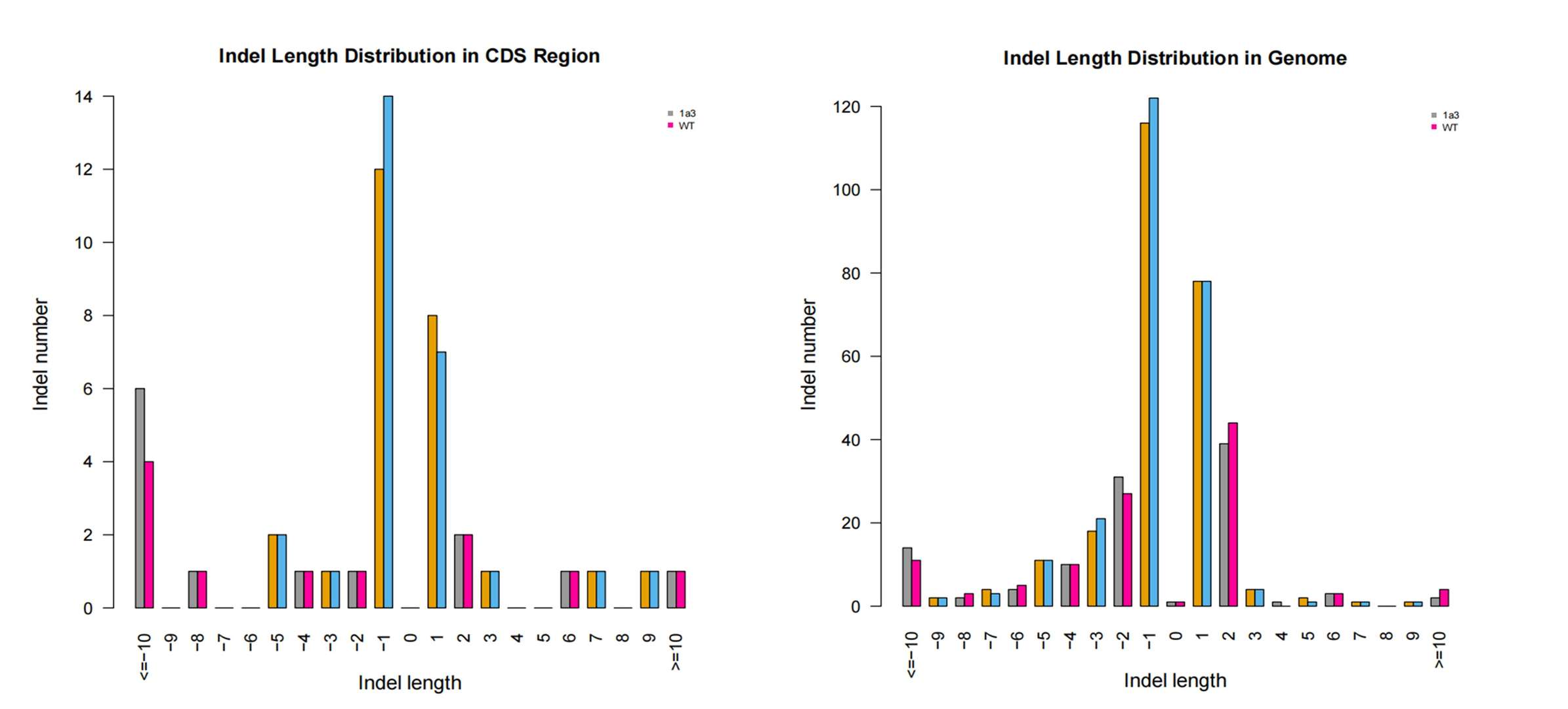
InDel-Längenverteilung sowohl im gesamten Genom als auch in den CDS-Regionen.
Statistik-Kreisdiagramm der InDel-Anmerkungen.
Häufig gestellte Fragen (FAQs)
Was ist die de novo Sequenzierung des gesamten Genoms von Pflanzen oder Tieren?
Es ist ein referenzfreier Ansatz zur Rekonstruktion des gesamten Genoms einer Art von Grund auf. Diese Methode ist entscheidend für Arten, die über ein zuverlässiges Referenzgenom verfügen oder solche mit komplexen strukturellen Variationen.
Wann sollte ich die de novo Genomsequenzierung anstelle der Resequenzierung wählen?
Antwort:
Wählen Sie de novo Sequenzierung, wenn:
- Es existiert kein hochqualitatives Referenzgenom.
- Ihre Spezies weist eine erhebliche genomische Vielfalt oder Komplexität auf.
- Sie zielen darauf ab, ein Pan-Genom zu erstellen oder die aktuelle Referenzqualität zu verbessern.
Welche Sequenzierungsplattformen werden in Ihrem Service verwendet?
Wir verwenden eine hybride Strategie, die kombiniert:
- Illumina (für k-mer-basierte Umfragen und Politur)
- PacBio HiFi / Oxford Nanopore (zur Erzeugung von Long-Read-Contigs)
- Hi-C (für Chromosomen-niveau Gerüstbildung)
Dieser schichtweise Ansatz maximiert die Kontinuität und Genauigkeit der Montage.
Welche Probenqualität wird benötigt?
Typische Anforderungen umfassen:
- Hochmolekulare gDNA
- OD260/280 = 1,8–2,0
- OD260/230 ≥ 2,0
- ≥10–15 μg Gesamt-DNA je nach Plattform
Wir stellen bei Anfrage detaillierte Einreichungsrichtlinien zur Verfügung.
Welche Ergebnisse werde ich erhalten?
Liefergegenstände umfassen:
- Hochwertig montiertes Genom (FASTA)
- Montagemetriken und Qualitätsbericht
- Genvorhersage- und funktionale Annotierungsdateien
- Visuelle Zusammenfassungen (z. B. Genomkreisdiagramme, Synteniekarten)
Bieten Sie eine nachgelagerte Analyse an?
Ja. CD Genomics bietet fortschrittliche bioinformatische Optionen, einschließlich:
- Orthologe Clustering
- Phylogenetische Rekonstruktion
- Analyse der Genfamilienexpansion
- Bewertung der Genom-Syntenie und Kollinearität
Kundenveröffentlichungshighlight
Fallstudie: Entschlüsselung der m6A-Methylierungsmechanismen in Arabidopsis mittels Whole-Genome de novo Sequenzierung
Journal: Neue Phytologe
Impact-Faktor8,3
Veröffentlicht: 2017
DOI: 10.1111/nph.14586
Hintergrund
Als Modellorganismus für die PflanzenGenetik, Arabidopsis thaliana war entscheidend für die Aufdeckung epigenetischer Regulationsmechanismen. Unter diesen, N6-Methyladenosin (m6A) Die Modifikation von mRNA spielt eine entscheidende Rolle im Wachstum, in der Entwicklung und in den Stressreaktionen von Pflanzen. Die molekularen Komponenten, die diese Modifikation antreiben – und ihre funktionale Erhaltung in höheren Pflanzen – sind jedoch noch unvollständig verstanden.
Diese Studie hatte zum Ziel, die genetischen Faktoren zu identifizieren, die für m6A-RNA-Methylierung in Arabidopsis durch Integration Whole-Genome-De-Novo-Sequenzierung mit gezielte funktionelle GenomikEin zentraler Fokus lag auf dem Verständnis der Rolle von HAKAI, einem konservierten E3 Ubiquitin-Ligaseinnerhalb der Methylierungsmaschinerie.
Materialien & Methoden
Genomanalyse und Mutantenscreening:
- Arabidopsis thaliana Mutanten mit vermuteten m6A-Methylierungsdefekten wurden aus T-DNA-Einfügungsbibliotheken ausgewählt.
- Genomisches DNA wurde extrahiert und de novo mit den Illumina-Short-Read- und ONT-Long-Read-Plattformen sequenziert, wobei eine hohe Kontinuität und Abdeckung erreicht wurde.
- Genomstörungen wurden kartiert und validiert.
m6A-Profilierung:
- Totale RNA wurde aus mutierten und Wildtyp-Linien isoliert.
- Die Quantifizierung von m6A wurde mittels LC-MS/MS und immunpräzipitationsbasiertem m6A-seq durchgeführt.
Funktionale Validierung:
- Komplementationstests wurden verwendet, um die Genfunktion zu überprüfen.
- RNA-Seq wurde angewendet, um die transkriptomischen Folgen des Verlusts von HAKAI zu bewerten.
Ergebnisse
Die gesamte de-novo-Sequenzierung des Genoms ermöglichte die genaue Identifizierung von T-DNA-Insertionsstellen, die stören. HAKAI, ein Gen, das eine RING-Domänen E3 Ubiquitin-Ligase kodiert. Der Funktionsverlust von HAKAI reduzierte die globalen m6A-Methylierungslevels erheblich, vergleichbar mit Mutanten von bekannte m6A-Schreiber wie zum Beispiel MTA und FIP37.
Wichtigste Erkenntnisse:
- Der Verlust von HAKAI führte zu Defekten in der apikalen Dominanz, der Blütezeit und der Embryolebensfähigkeit, was die Phänotypen anderer Kern-m6A-Komponentenmutanten nachahmte.
- Die Transkriptomanalyse zeigte eine Dysregulation in wichtigen Entwicklungs- und Hormonsignalwegen.
- Die Komplementation des HAKAI-Gens stellte sowohl die m6A-Methylierungsniveaus als auch die normale Entwicklung wieder her.
Fazit
Diese Studie hat gezeigt, dass HAKAI ist ein kritischer Bestandteil des m6A-Methylierungs-Komplexes. In Pflanzen wirken sie neben den kanonischen Methyltransferasen. Die Verwendung von Whole-Genome-De-Novo-Sequenzierung ermöglichte eine präzise Kartierung von Genunterbrechungen und war entscheidend für die Validierung funktionaler Hypothesen in genetisch komplexen Hintergründen.
Der Fall hebt hervor, wie Pflanzen-Whole-Genome-De-Novo-Sequenzierung, gepaart mit epitranskriptomischen und transkriptomischen Werkzeugen, kann konservierte regulatorische Mechanismen aufdecken. CD Genomics unterstützt ähnliche Studien, indem es integrierte Genomassemblierung, Methylierungsanalyseund funktionelle Genomik Pipelines für Pflanzenepigenetik und darüber hinaus.
Hier sind einige Veröffentlichungen, die erfolgreich mit unseren Dienstleistungen oder anderen verwandten Dienstleistungen veröffentlicht wurden:
Kombinationen von Bakteriophagen sind wirksam gegen multiresistente Bakterien. Pseudomonas aeruginosa und die Empfindlichkeit gegenüber Carbapenem-Antibiotika erhöhen
Journal: Viren
Jahr2024
Genomsequenz, Antibiotikaresistenzgene und Plasmide in einer monophasischen Variante von Salmonella typhimurium isoliert von Einzelhandelsfleisch vom Schwein
Tagebuch Ankündigungen zu Mikrobiologie-Ressourcen
Jahr2024
Gene von Salmonella enterica Serovar Enteritidis, das an der Biofilmbildung beteiligt ist
Journal: Angewandte Mikrobiologie
Jahr2024
Vollständige Genomsequenz des Probiotikums Bifidobacterium adolescentis Stamm iVS-1
Journal: Ankündigungen zu Mikrobiologie-Ressourcen
Jahr: 2023
Mehr anzeigen Artikel, die von unseren Kunden veröffentlicht wurden.


 Richtlinien zur Einreichung von Proben
Richtlinien zur Einreichung von Proben